-
TP dissection patte + dessins à légender
dissection en video : http://youtu.be/u1qfg9LehzM
Schéma patte : http://pedagogie.ac-toulouse.fr/sciences31/cahierdexperiences/cahierscan/mouvement_corps/original/corps05.jpg
fiche TP à télécharger : http://www.cndp.fr/ecole/sciences/classeurs/fiches_eleves/ch_word/ch60.doc
schéma bras http://www.trikapalanet-fr.com/wp-content/uploads/2009/12/flexionextension.jpg ; http://www.docteurclic.com/photos/muscles_contraction.jpg ;
schéma imprimable (gif) : http://pst.perso.libertysurf.fr/svtiufm/crpe/crpe1996.htm
 votre commentaire
votre commentaire
-
http://www2.cnrs.fr/presse/communique/2621.htm
Le mimétisme est un phénomène très répandu dans la nature : nombreuses sont les espèces qui s'imitent les unes les autres dans leur apparence afin de mieux se défendre des prédateurs. Un consortium international impliquant des chercheurs du CNRS/MNHN (laboratoire Origine, Structure et Evolution de la Biodiversité) et de l'INRA (Physiologie de l'insecte : communication et signalisation) vient pour la première fois de séquencer et d'assembler le génome complet du papillon tropical Heliconius melpomene. Grâce à ce génome de référence, les chercheurs montrent que la ressemblance mimétique est rendue possible grâce à l'échange des gènes de la couleur entre espèces différentes. Jusqu'à présent, les hybridations entre espèces voisines étaient vues comme néfastes, car produisant des descendants généralement moins compétitifs et peu performants. En réalité, elles permettent aussi le transfert de gènes offrant un avantage sélectif, ici la marque colorée de la toxicité de ces papillons pour leurs prédateurs. Ces résultats sont publiés le 16 mai 2012 sur le site de la revue Nature.
Heliconius est un genre de papillon comptant une cinquantaine d'espèces présentes dans les forêts d'Amérique Centrale et du Sud. Pour les chercheurs, il constitue un modèle de choix pour étudier la sélection naturelle et les changements génétiques qui mènent à la formation de nouvelles espèces. Afin de pousser plus loin ces travaux et d'avoir une vue plus large des mécanismes génétiques en œuvre, un consortium international d'équipes de recherche a réalisé le séquençage du génome de l'un de ces lépidoptères, Heliconius melpomene, originaire du Panama. Désormais, l'ensemble du génome de ce papillon, incluant plus de 12 600 gènes répartis sur 21 chromosomes, est accessible. Si, à ce jour, relativement peu de génomes d'insectes ont été séquencés, il est encore plus rare de disposer de celui d'une espèce non domestiquée telle que Heliconius melpomene.
Ce séquençage a permis d'obtenir des résultats étonnants sur le mimétisme. Il était connu des chercheurs que H. melpomene partage les motifs et les couleurs d'une espèce voisine, H. timareta. Ces deux espèces sont toxiques et les couleurs vives de leurs ailes leur permettent d'être facilement reconnues des prédateurs. En adoptant la même coloration, elles partagent le coût que représente « l'éducation » de ces derniers. En effet, c'est en mangeant quelques Heliconius que les prédateurs, des oiseaux pour la plupart, apprennent à associer les couleurs au mauvais goût et à la toxicité de ces insectes. De ce fait, la ressemblance des deux espèces constitue pour elles un important avantage sélectif.
Jusqu'à présent, on pensait que ce mimétisme était dû à une convergence génétique : chaque espèce devait développer ses propres innovations génétiques permettant de ressembler à sa voisine. Mais le séquençage du génome de H. melpomene vient de démontrer que la ressemblance visuelle entre cette espèce et H. timareta est due à des accouplements croisés entre ces deux papillons. En effet, les chercheurs ont découvert que, pour ces deux espèces, les régions du génome qui contrôlent la couleur des ailes ont une origine unique et récente. Ces gènes sont donc passés de l'une à l'autre par hybridation.
Les accouplements entre ces deux espèces sont très rares et seule la descendance mâle est fertile. Néanmoins, ils sont suffisants pour transmettre à l'espèce voisine les traits phénotypiques qui confèrent la ressemblance. Ces résultats mettent à mal le paradigme selon lequel l'hybridation entre espèces est forcément néfaste car elle produit des individus fragiles ou mal adaptés aux niches écologiques des espèces parentes. Au contraire, l'hybridation permet ici le passage d'innovations génétiques « prêtes à l'emploi » d'une espèce à l'autre, et constitue ainsi un moteur de l'évolution à ne pas négliger. Ces résultats pourraient s'appliquer à d'autres adaptations comme la résistance chez certains insectes tels que les moustiques Anopheles, les traits de domestication ou encore, les gènes d'immunité du génome humain.
Parallèlement à l'étude du mimétisme, le séquençage du génome de H. melpomene a permis de décrire certaines fonctions peu connues chez ces lépidoptères, et là aussi les résultats bouleversent les idées reçues. On pensait notamment que les papillons diurnes, à grande acuité visuelle, avaient une communication olfactive moins développée que les papillons de nuit. Or, les chercheurs ont montré qu'au contraire, H. melpomene possède une grande diversité de récepteurs olfactifs, comparable à celle des papillons nocturnes, tel le bombyx du mûrier (ver à soie), et même des expansions de certaines familles de gènes chimiosensoriels. Ceci ouvre un vaste champ de recherche sur la complexité de la communication chimique des papillons de jour.

© Chris Jiggins, University of Cambridge. Cette image est disponible à la photothèque du CNRS, phototheque@cnrs-bellevue.fr
Heliconius melpomene, dont le génome est à présent entièrement séquencé.

© Chris Jiggins, Cambridge, 2009. Cette image est disponible à la photothèque du CNRS, phototheque@cnrs-bellevue.fr
Le papillon Heliconius melpomene, dont le génome est à présent entièrement séquencé.
Les motifs colorés de ses ailes sont utilisés comme signaux de leur toxicité, et leur permettent d'être évités par les prédateurs avertis.
Références :
The Heliconius Genome Consortium. "Butterfly genome reveals promiscuous exchange of mimicry adaptations between species". Nature, DOI: 10.1038/nature11041. Publication le 16 mai 2012.
 votre commentaire
votre commentaire
-
http://www2.cnrs.fr/presse/communique/2629.htm
Des équipes de chercheurs de l'Institut Pasteur et du CNRS viennent de mettre au point, en combinant deux méthodes récentes d'imagerie, une nouvelle approche de microscopie optique permettant de visualiser des assemblages moléculaires avec une résolution environ 10 fois meilleure que les microscopes traditionnels, tout en respectant leur fonction biologique. Grâce à cette approche, ils ont pu observer, pour la première fois dans une cellule par voie optique, le virus du sida et sa capside (contenant le génome du VIH) à une résolution de 30 nanomètres. L'approche développée représente une avancée majeure, ouvrant la voie à des analyses moins invasives et plus précises de micro-organismes pathogènes présents dans des cellules hôtes humaines vivantes. Cette étude est en ligne sur le site de la revue PNAS.
Depuis toujours, il est nécessaire pour les chercheurs de pouvoir visualiser les virus qu'ils étudient dans l'environnement de leur cellule cible, afin de définir les interactions hôte-pathogène contribuant à l'infection. La microscopie optique, qui utilise des molécules fluorescentes (comme les protéines GFP ou des anticorps couplés à des fluorophores synthétiques) permet de mettre en avant les différentes structures d'une cellule, dont les protéines. Cependant, cette méthode est limitée par son faible pouvoir de résolution, ne pouvant distinguer des structures cellulaires et moléculaires qu'à une échelle de 200 à 300 nanomètres (nm). La plupart des virus étant de taille inférieure, il est nécessaire de recourir à des techniques d'imagerie plus précises, afin de définir leur structure interne.
Une étude coordonnée par le Dr Christophe Zimmer(1) (Institut Pasteur/CNRS), en collaboration avec le Dr Nathalie Arhel(2) au sein du laboratoire du Pr Pierre Charneau(3) (Institut Pasteur/CNRS), révèle que l'association de deux techniques récentes d'imagerie permet d'obtenir des images uniques d'assemblages moléculaires de la capside du VIH-1, avec une résolution environ 10 fois meilleure que les microscopes optiques traditionnels. Cette approche, qui utilise la microscopie super-résolutive PALM et le marquage FlAsH, n'affecte pas la capacité du virus à se répliquer. Elle représente une avancée majeure pour la recherche en biologie moléculaire, permettant de visualiser des complexes microbiens à une échelle de 30 nm dans les cellules sans perturber leur fonction.
L'approche développée combine la microscopie super-résolutive PALM et le marquage FlAsH. La microscopie PALM se fonde sur la prise de milliers de clichés en basse résolution, dont chacun ne montre que quelques molécules fluorescentes. Les positions de ces molécules sont ensuite calculées et assemblées par ordinateur afin d'obtenir une seule image en haute résolution. Le marquage FlAsH, quant à lui, implique la fusion d'un peptide de 6 acides aminés à la protéine étudiée, auquel se lie le fluorophore FlAsH. Cette liaison génère de la fluorescence, permettant ainsi la visualisation de cette protéine. C'est la première fois qu'une équipe de chercheurs regroupe ces deux méthodes afin d'obtenir des images en haute-définition d'une structure moléculaire aussi bien dans des cellules fixées que dans des cellules vivantes.
Grâce à cette nouvelle approche, les chercheurs ont pu visualiser la morphologie du virus du sida à une échelle de 30 nm et localiser sa capside dans des cellules humaines. Les capsides sont des structures coniques qui contiennent le génome du VIH. Ces structures doivent se défaire pour permettre au génome du VIH de s'intégrer dans celui de la cellule hôte. Cependant, la chronologie de ce désassemblage a longtemps été débattue. Selon une hypothèse dominante, la capside se désassemblerait immédiatement après infection de la cellule et ne jouerait donc qu'un rôle marginal dans le voyage intracellulaire du virus vers le noyau. Les résultats obtenus par les chercheurs de l'Institut Pasteur et du CNRS indiquent, au contraire, que de nombreuses capsides restent intactes jusqu'à l'entrée du VIH dans le noyau des cellules, confirmant et renforçant de précédentes études en microscopie électronique. Ainsi, les capsides pourraient jouer un rôle plus important que communément admis dans le cycle réplicatif du virus.
Le développement de cette nouvelle approche de microscopie optique par les chercheurs de l'Institut Pasteur et du CNRS offre des perspectives uniques pour la biologie moléculaire. En effet, cette nouvelle technique d'imagerie pourrait devenir un outil important dans l'analyse de nombreux complexes microbiens et de leurs interactions avec des cellules hôtes à l'échelle moléculaire. Cette technique non-invasive permet d'observer des protéines sans détruire, ni altérer, leur fonction biologique. Par ailleurs, elle pourrait, à terme, rendre possible l'analyse de micro-organismes à des résolutions de l'ordre du nanomètre, permettant ainsi de passer de la microscopie à la « nanoscopie ». La prochaine étape sera, par conséquent, le partage de cette approche avec la communauté scientifique, son développement et son application à l'étude d'autres micro-organismes pathogènes.
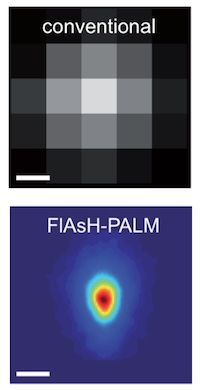
© Institut Pasteur
Reconstruction optique super-résolutive de la morphologie du VIH. L'image du dessous montre la distribution moyenne de l'enzyme intégrase observée par FlAsH-PALM. La résolution de cette technique (~30 nm) permet de retrouver la taille et la forme conique de la capside. Pour comparaison, la résolution de la microscopie conventionnelle (~200-300 nm), illustrée par l'image du dessus, ne permet pas une description détaillée de cette structure.
Notes :
(1) Dr Christophe Zimmer, chef du groupe Imagerie et Modélisation (Institut Pasteur) ; CNRS URA 2582
(2) Dr Nathalie Arhel, unité Virologie moléculaire et Vaccinologie (Institut Pasteur) ; CNRS URA 3015
(3) Pr Pierre Charneau, chef de l'unité Virologie moléculaire et Vaccinologie (Institut Pasteur) ; CNRS URA 3015Références :
Super resolution imaging of HIV in infected cells with FlAsH-PALM, en ligne sur le site de PNAS, le 14 mai 2012. Mickaël Lelek, Ricardo Henriques et Christophe Zimmer : Institut Pasteur, Groupe Imagerie et Modélisation, CNRS URA 2582, Paris. Francesca Di Nunzio, Pierre Charneau et Nathalie Arhel : Institut Pasteur, Molecular Virology and Vaccinology Unit, CNRS URA 3015, Paris.
 votre commentaire
votre commentaire
-
A2/ Expérience sur cœur d’huître /video
http://www.snv.jussieu.fr/bmedia/ATP/dis-hui.htm
http://svt.ac-dijon.fr/schemassvt/article.php3?id_article=686
automatisme cardiaque
 votre commentaire
votre commentaire
-
Test circulation-respiration
Correction devoir commun
Ière Partie : restitution organisée des connaissances /10 points
-
intro + concl : en 2 parties au moins
-
plan cohérent + titres soulignés
-
schéma coupe de sol soigné légendé + titre
-
+ litière + horizons + biosphère + roche mère
-
définition du sol : entre bio et lithosphère, horizons humifère et d'accumulation ou altération
-
lenteur formation, différents sols, variation épaisseur, composition, exemples
-
formation du sol / décomposition roche mère / + altération physique + chimique / eau + végétaux
-
formation du sol / décomposition matière organique / décomposeurs
-
dégradation sol / facteurs chimiques, érosion, température
-
dégradation sol / facteurs humains : déforestation, labour, pollution, tassement, urbanisation
IIème Partie : exploitation de documents / 10 points
Exercice 1 Hachette n°13p.158 / 4+2 = 6 points
-
histogramme
-
titre
-
légende axes + unités
-
échelle et mise en page judicieuse, logique
-
baisse du charbon + gaz depuis 1973, pétrole depuis 1990, du nucléaire depuis 2006, augmentation éolien depuis 1973
-
remplacement énergie fossile / éolienne + solaire + hydrolique
Exercice 2 Nathan n°12p.169 / 2+2 = 4 points
-
avantage culture hors sol des algues par rapport au maïs ou canne : pas d'utilisation du sol
-
" 100 kg d'algues ont absorbé 182 kg de CO2 ".
-
pas d'utilisation d'énergie fossile, donc de pb de réserves disponibles
-
moins d'effet sur l'évolution du taux de CO2. " atténuer son empreinte carbone "
 votre commentaire
votre commentaire
-
Mon petit cahier de sciences naturelles





