-
http://www2.cnrs.fr/presse/communique/2336.htm
Les premiers Hommes modernes (Homo sapiens) seraient arrivés en Europe il y a environ 45 000 ans, soit plusieurs millénaires avant la date communément admise jusqu'ici. Fruit d'une collaboration entre treize équipes européennes, à laquelle participent deux chercheurs français du CNRS et de l'Université Bordeaux 1 (1), ce résultat s'appuie sur de nouvelles analyses de deux dents de lait découvertes il y a une cinquantaine d'années dans une grotte préhistorique italienne, et qui avaient été attribuées à tort à des Néanderthaliens. Ces restes humains, datant d'il y a environ 45 000 ans, s'avèrent appartenir à Homo sapiens. Ils constituent les plus anciens témoignages d'Hommes modernes européens connus à ce jour. Publiés le 3 novembre dans la revue Nature, ces travaux apportent de nouveaux éléments pour mieux comprendre la diffusion des premiers Hommes modernes en Europe, ainsi que la période dite de « transition », allant de leur arrivée en Europe à la disparition des Néanderthaliens.
Selon l'hypothèse la plus largement partagée à ce jour, la disparition de l'Homme de Néanderthal, qui a vécu en Europe pendant plus de 200 000 ans, aurait un lien avec l'arrivée sur ce même continent des Hommes anatomiquement modernes (Homo Sapiens). Encore largement débattue dans la communauté scientifique, la question complexe de leur extinction vient de recevoir de nouveaux éléments de réflexion, grâce à une collaboration scientifique européenne qui s'est intéressée à deux dents de lait retrouvées par M. Palma di Cesnola (Université de Sienne), dans la Grotta del Cavallo, située près de la petite ville d'Uluzzo, au sud de l'Italie. Découverte en 1960, cette grotte contient des dépôts archéologiques témoignant de la période pendant laquelle les Néanderthaliens ont été remplacés par les Hommes modernes. Décrite à partir de plus de vingt sites archéologiques en Italie, la culture « uluzzienne » est caractérisée par la présence d'objets (ornements personnels, outils en os, colorants, etc…) typiquement associés à un comportement symbolique des Hommes modernes. Or, lors de précédents travaux, les dents de Cavallo furent attribuées aux Néanderthaliens. Ces derniers ont alors été considérés comme les artisans des ornements et des outillages caractéristiques de la culture « uluzzienne ».
De nouvelles analyses effectuées par une équipe internationale impliquant deux laboratoires français viennent contredire ces précédentes conclusions. Les reconstructions en 3D, issues d'enregistrements par microtomographie RX (2), des restes humains de Cavallo ont été comparées à un large échantillon de dents néanderthaliennes et modernes. En analysant les paramètres de leur structure interne et externe (en particulier l'épaisseur de l'émail et le contour des couronnes (3) dentaires), les chercheurs ont mis en évidence que les deux dents de Cavallo appartenaient à des Hommes modernes. D'autre part, la datation au carbone 14 par méthode AMS (4) sur des coquilles perforées, issues des mêmes niveaux archéologiques que les dents, a montré que ce matériel serait vieux d'environ 43 000 à 45 000 ans.
Ces résultats indiquent une arrivée plus précoce d'Homo sapiens en Europe. Ils confirment la longue période de coexistence des Hommes modernes avec les Néanderthaliens. De plus, cette étude suggère que, contrairement à ce qui a été affirmé par le passé, les Hommes modernes seraient les artisans de la culture uluzzienne. Cette découverte apporte de nouvelles données pour comprendre le développement des comportements symboliques des populations du Paléolithique. Issue de la réévaluation des deux dents de Cavallo, elle n'aurait pas été possible sans une collaboration entre plusieurs institutions européennes et le recours aux innovations techniques développées au cours de la dernière décennie.

© Annamaria Ronchitelli
La Grotta del Cavallo (flèche rouge) s'ouvre sur la baie d'Uluzzo, située dans le Parc Régional Naturel de Portoselvaggio (Pouilles, sud de l'Italie).

© Annamaria Ronchitelli et Dr. Katerina Douka
Artefacts uluzziens de la Grotta del Cavallo (Pouilles, sud de l'Italie).

© Dr. Stefano Benazzi.
Vue mésiale du spécimen Cavallo-B (première molaire déciduale supérieure gauche), le premier Homme anatomiquement moderne d'Europe. La barre blanche dans la figure est équivalente à 1 cm.
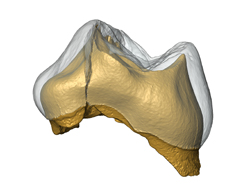
© Dr. Stefano Benazzi.
Reconstruction numérique en 3D de Cavallo-B (première molaire déciduale supérieure gauche). L'émail est en transparence pour montrer la dentine de la couronne.
Notes :
(1) Unité « Anthropologie bioculturelle » (CNRS / Université de la Méditerranée /EFS Alpes Méditerranée) qui s'intéresse d'un point de vue paléoanthropologique à la problématique de la « transition » entre les Néanderthaliens et les Hommes modernes et Unité « De la Préhistoire à l'actuel : culture, environnement et anthropologie » (CNRS / Université Bordeaux 1 / ministère de la Culture et de la Communication) qui a mis à disposition de cette étude ses compétences et ses équipements en matière d'imagerie 3D à haute résolution appliquée à la paléoanthropologie.
(2) Scanner CT (Computed Tomography) à haute résolution.
(3) Partie de la dent recouverte d'émail, en opposition à la racine.
(4) Méthode AMS ou en français SMA : datation par "spectrométrie de masse par accélérateur".
Références :
Early dispersal of modern humans in Europe and implications for Neanderthal behaviour, Stefano Benazzi, Katerina Douka, Cinzia Fornai, Catherine C. Bauer, Ottmar Kullmer, Jiri Svoboda, Ildiko Pap, Francesco Mallegni, Priscilla Bayle, Michael Coquerelle, Silvana Condemi, Annamaria Ronchitelli, Katerina Harvati & Gerhard W.Weber, Nature, 3 novembre 2011
 votre commentaire
votre commentaire
-
http://www2.cnrs.fr/presse/communique/2335.htm
L'équipe AVENIR Inserm "Plasticité génomique et vieillissement" dirigée par Jean Marc Lemaitre, chargé de recherche Inserm à l'Institut de génomique fonctionnelle (Inserm/CNRS/Université de Montpellier 1 et 2), vient de parvenir à rajeunir des cellules de donneurs âgés, vieilles de plus de 100 ans. Ces cellules âgées, reprogrammées in vitro en cellules souches pluripotentes (iPSC pour "Induced pluripotent stem cells") ont retrouvé leur jeunesse et les caractéristiques des cellules souches embryonnaires (hESC): elles peuvent se différencier à nouveau en cellules de tous types après une véritable cure de "jouvence". Ces résultats constituent une avancée significative pour la recherche sur les cellules iPSC et une nouvelle étape vers la médecine régénérative.
Les résultats sont publiés dans la revue Genes & Development datée du 1er novembre 2011.Les cellules souches embryonnaires humaines (hESC) sont des cellules "à tout faire" qui sont indifférenciées. Par leurs divisions, elles assurent la mise en place de toutes les cellules adultes différenciées de l'organisme (neurones, cellules cardiaques, cellules de peau, cellules du foie…cf. figure 1).
Depuis 2007, quelques équipes de recherche dans le monde sont capables de reprogrammer des cellules adultes humaines en cellules souches pluripotentes (iPSC), qui présentent des caractéristiques et un potentiel similaires aux cellules souches embryonnaires humaines (hESC). Cette reprogrammation (cf. figure 1 en rouge), offre la possibilité de reformer tous les types cellulaires de l'organisme en dehors des contraintes éthiques liées à l'utilisation de cellules souches de type embryonnaires.
Jusqu'alors, les résultats de recherches publiés montraient que la sénescence, point ultime du vieillissement cellulaire, restait une limite à l'utilisation de cette technique pour des applications thérapeutiques chez des patients âgés.
Aujourd'hui, Jean Marc Lemaitre, chargé de recherche à l'Inserm et son équipe, viennent de franchir cette limite. Les chercheurs sont parvenus à rajeunir des cellules de donneurs âgés, jusqu'à plus de 100 ans, et ont ainsi démontré la réversibilité du processus du vieillissement cellulaire.
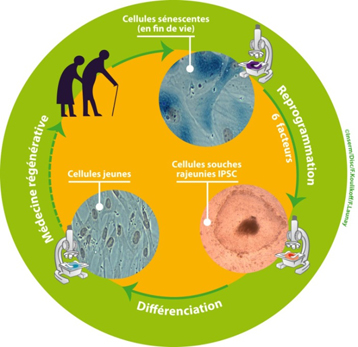
Pour ce faire, ils ont utilisé une stratégie adaptée qui consiste à reprogrammer des cellules, grâce à un "cocktail" spécifique de 6 facteurs génétiques tout en effaçant les marques du vieillissement. Les chercheurs ont montré que les cellules souches iPSC obtenues ont alors la capacité de reformer tous les types cellulaires de l'organisme. Elles possèdent des caractéristiques physiologiques de cellules "jeunes", tant du point de vue de leur capacité proliférative que de leur métabolisme cellulaire.
Un cocktail de 6 facteurs génétiques…
Les chercheurs ont d'abord multiplié des cellules de la peau (fibroblastes) d'un donneur de 74 ans pour atteindre la sénescence caractérisée par l'arrêt de la prolifération des cellules. Ils ont ensuite procédé à la reprogrammation in vitro de ces cellules. Dans cette étude, Jean Marc Lemaitre et son équipe ont d'abord confirmé que cela n'était pas possible avec le lot de 4 facteurs génétiques classiquement utilisé (OCT4, SOX2, C MYC et KLF4) et ont ajouté 2 facteurs supplémentaires (NANOG et LIN28) qui ont permis de franchir cette barrière (cf. figure 2).
Grâce à ce nouveau "cocktail" de 6 facteurs, les cellules sénescentes, reprogrammées en cellules souches pluripotentes iPSC fonctionnelles, réacquièrent les caractéristiques de cellules souches pluripotentes de type embryonnaire.
En détail, elles ont retrouvé leur capacité d'autorenouvellement et leur potentiel de différenciation d'antan, ne conservant aucune trace de leur vieillissement antérieur.
Pour vérifier les caractéristiques "rajeunies" de ces cellules, les chercheurs ont testé le processus inverse. Les cellules iPSC rajeunies ont été à nouveau différenciées en cellules adultes (cf. figure 1) et comparées aux cellules âgées d'origine ainsi qu'à celles obtenues à partir de cellules souches pluripotentes de type embryonnaires (hESC).
"Les marqueurs de l'âge des cellules ont été effacés et les iPSC, que nous avons obtenues peuvent produire des cellules fonctionnelles, de tous types avec une capacité de prolifération et une longévité accrues", explique Jean Marc Lemaitre qui dirige l'équipe AVENIR de l'Inserm.
...testé sur des cellules âgées de plus de 100 ans
Les résultats obtenus ont conduit l'équipe de recherche à tester le cocktail sur des cellules plus âgées de 92, 94, 96 jusqu'à 101 ans. "Notre stratégie a fonctionné sur les cellules de centenaires. L'âge des cellules n'est définitivement pas une barrière à la reprogrammation", conclut-il. "Ces travaux ouvrent la voie à l'utilisation thérapeutique des iPS à terme, en tant que source idéale de cellules adultes tolérées par le système immunitaire, pour réparer des organes ou des tissus chez des patients âgés", ajoute le chercheur.
Ce travail a fait l'objet d'une demande de brevet auprès d'Inserm Transfert.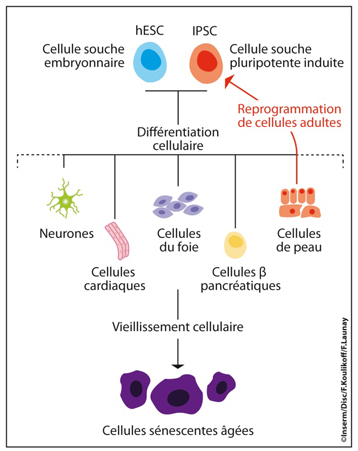
© Inserm/Disc/F.Koulikoff/F.Launay
Figure 1 : Différenciation cellulaire
© Inserm/Disc/F.Koulikoff/F.Launay
Figure 2 : Reprogrammation de cellules sénescentes âgées
Références :
Rejuvenating senescent and centenarian human cells by reprogramming through the pluripotent state
Lapasset, L. (1), Milhavet, O. (2), Prieur, A. (1), Besnard, E. (1), Babled, A. (1), Aït-Hamou, N. (1), Leschik, J. (1), Pellestor, F. (4), Ramirez, JM. (3), De Vos, J. (3), Lehmann, S. (2), Jean-Marc Lemaitre (1).
1- Laboratory of Plasticity of the Genome and Aging, Institute of Functional Genomics, 141 rue de
la Cardonille, 34094 Montpellier Cedex 05, France
2- Laboratory of Neurological Disorders and Stem Cells, Institute of Human Genetics, 141 rue de
la Cardonille, 34396 Montpellier Cedex 05, France
3- Laboratory of Early Embryo Development and Human Pluripotent Stem Cells, Institute for
Research in Biotherapy, CHU Saint-Eloi Hospital, 80 avenue Augustin Fliche, 34295 Montpellier
Cedex 05, France
4- Laboratory of chromosomal Genetics, Medical Genetics Department Hôpital CHRU Arnaud de
Villeneuve, 371 avenue du Doyen Gaston Giraud, 34295 Montpellier cedex 5, France
Genes & Development, 1er novembre 2011. Vol. 25, No. 21, doi:10.1101/gad.173922.111 votre commentaire
votre commentaire
-
Allèles = versions d'un gène
Gène = information génétique = plan de fabrication d'une protéine, codé dans l'ADN par la séquence des bases, transmis de génération en génération.
Génôme = programme génétique = ensemble des gènes d'une cellule, d'un individu
A4/ Schématisation d'une transgénèse
une souris verte, ...fluo : http://tubeaessai.blogs.nouvelobs.com/archive/2008/01/index.html ;http://www.greenpeace.org/canada/fr/Blog/cchons-ogm-vert-fluo-dune-mduse/blog/4029 ;http://www.bio.miami.edu/~cmallery/255/255hist/greenmice.jpg ; http://genetique.snv.jussieu.fr/BMC513.htm ;http://www.mbl.edu/news/features/shimomura.html ;http://www.collegeotr.com/boston_university/ucsd_bu_and_columbia_scientists_bring_home_nobel_prize_in_chemistry_make_stuff_glow_12809; http://www.scientificamerican.com/blog/post.cfm?id=chalfie-shimomura-and-tsien-win-che-2008-10-08
=> transgenesis, DNA genes support and universal
[OGM, transgénèse, ADN support gènes et universel]
A5/ Débat sur éthique, clonage, transgénèse et ogm
TP clonage : http://www.emilangues.education.fr/ressources-pedagogiques/sequences/disciplines-non-linguistiques/adn&page=sequence
un article sur le clonage animal : http://biotechbuddy.googlepages.com/animal_cloning.html
l'article de Wikipédia sur Dolly en anglais : http://en.wikipedia.org/wiki/Dolly_the_Sheep
la page de wikipédia sur le clonage en anglais : http://en.wikipedia.org/wiki/Cloning
les Églises d’Écosse donnent leur point de vue éthique sur le clonage : http://www.srtp.org.uk/srtpage3.shtml
des articles d'actualité sur le clonage : http://news.bbc.co.uk/2/hi/science/nature/377046.stm
un article sur le clonage d'un faon au Texas : http://www.innovations-report.de/html/berichte/biowissenschaften_chemie/bericht-
24409.html
des vidéos qui expliquent le clonage : http://www.hhmi.org/biointeractive/stemcells/animations.html#scnt_anim
La carte du maïs OGM en France : http://www.futura-sciences.com/fr/sinformer/actualites/news/t/genetique-1/d/la-carte-du-mais-ogm-en-france_12336/
MON 863 : maïs OGM destiné à l'alimentation humaine suspecté puis innocenté : http://www.futura-sciences.com/fr/sinformer/actualites/news/t/genetique-1/d/mon-863-mais-ogm-destine-a-lalimentation-humaine-suspecte-puis-innocente_12332/
films sur les OGM :
utilisé comme sujet choc : http://www.hoaxbuster.com/hoaxliste/hoax.php?idArticle=54669
de journalistes :
http://www.dailymotion.com/video/xrn35_ogm-lhorreur-reveillez-vous-avant
http://www.dailymotion.com/video/xqqe3_le-coton-ogm-tue
http://www.dailymotion.com/video/x119pg_main-basse-sur-les-genes
de scientifique, C.Vellot :
http://www.dailymotion.com/video/xpwco_eclairage-scientifique-sur-les-ogm
http://www.dailymotion.com/video/xpync_eclairage-scientifique-sur-les-ogm
 votre commentaire
votre commentaire
-
Rappels à partir de schbilans© à Compléter
® matrices©
322 – Groupes phylogénétiques
matrices-arbres©
construire arbre vertébrés avec phylo
=> état "primitif" = "ancestral" // "dérivé" = "évolué" d'un caractère.
Un caractère ancestral, ou primitif ne permet pas de regrouper des êtres vivants puisque cet état de caractère peut être partagé avec d'autres êtres vivants d'un autre groupe. Les êtres vivants ou fossiles qui partagent des états dérivés de caractères forment un ensemble appelé taxon, d'où le terme de taxonomie.
établir une phylogénie revient à regrouper les espèces qui partagent les mêmes états dérivés pour un ensemble de caractères donnés. On classe les espèces dans des groupes phyl(ogén)étiques de plus en plus restreints. un groupe monophylétique (= clade => cladistique) est l'ensemble des espèces partageant un même état dérivé (= innovation évolutive). Plus des espèces partagent d'états dérivé, plus elles sont proches, plus leur ancêtre commun est récent. ,
schéma-bilan p35
=> groupe mono, para, polyphylétique
 votre commentaire
votre commentaire
-
A5/ schématisation sur la synthèse des protéines
Dia synthese_proteines / dessin synthese_proteines © à compléter
http://fr.wikivisual.com/images/f/f3/Protéines.png
http://svt.ac-dijon.fr/schemassvt/IMG/gif/synpro2.gif
http://upload.wikimedia.org/wikipedia/commons/7/73/Transcription_traduction.png
http://www.ac-creteil.fr/biotechnologies/doc_biochemistry-dogme_central-test.htm
polypeptide, ARNpm, maturation ARNm, localisation,
brin codant de l’ADN
transcription, traduction,
phénomène
Transcription
Traduction
Réplication = duplication
plan de fabrication
ADN brin transcrit
ARNm brin codant
ADN entier
matière première
nucléotides AUGC
20 acides aminés
nucléotides ATGC
machine
ARN polymérase
Ribosome
ADN polymérase
produit
ARN
polypeptide
ADN entier
localisation
nucléoplasme
cytoplasme
nucléoplasme
 votre commentaire
votre commentaire
Mon petit cahier de sciences naturelles



